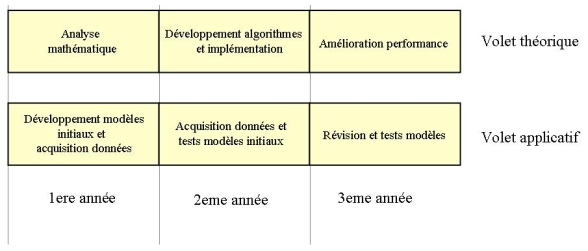
Au jour d'aujourd'hui (décembre 2004) le projet avance comme
prévu. Un article sur l'analyse mathématique des points
d'équilibre des équations différentielles LPM et de
leur stabilité a été soumis, alors qu'une
implémentation prototype d'un module de GNA pour l'identification
de points d'équilibre est actuellement en cours. Un
modèle initial de la régulation globale de la
transcription chez
Escherichia coli
a été construit et est décrit dans un autre article
soumis pour publication. Afin de tester les prédictions du
modèle, des expériences pour mesurer le profil
d'expression des gènes clés du modèle ont
été lancées. En outre, un premier modèle du
processus correspondant chez
Synechocystis
est sur le point d'être fini.
Plusieurs participants à ce projet sont également
impliqués dans le projet "
GDyn :
Analyse dynamique de réseaux de régulation génique",
financé dans le cadre des
Actions de
Recherche Coopérative (ARC) de l'
INRIA.
Pour nous contacter :